 |
연구 개요도 |
<이미지를 클릭하시면 크게 보실 수 있습니다> |
국내 연구진이 메신저 리보핵산(mRNA) 치료제의 품질을 좌우하는 핵심 구조인 'poly(A) 꼬리' 길이를 정밀하고 정확하게 분석할 수 있는 새로운 기술을 개발했다.
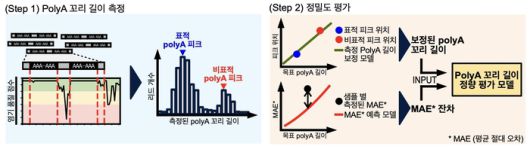
아주대학교(총장 최기주)는 박대찬 첨단바이오융합대학 교수와 남재환 가톨릭대 의생명과학과 교수 공동 연구팀이 mRNA 치료제의 poly(A) 꼬리(poly(A)denylation tail)를 정량적으로 분석하는 '3AIM-seq' 시퀀싱 기술을 최초로 개발했다고 21일 밝혔다.
poly(A) 꼬리는 mRNA의 3' 말단에 위치하며, 반복된 아데닌 염기가 줄지어 있는 구조다. 이 꼬리는 mRNA의 안정성과 단백질 발현 효율에 핵심적이며, mRNA 치료제의 의약학적 유효성과 안전성에 직접적인 영향을 미치는 것으로 알려져 있다.
하지만 poly(A) 꼬리는 수십~수백 개의 동일 염기가 반복돼 있어 기존 시퀀싱 방식으로는 삽입·삭제 오류가 잦아 정확한 품질 평가가 어려웠다. 특히 최근에는 중간에 링커(linker) 서열 등이 삽입된 구조적 변형이 꾸준히 개발되면서, 꼬리 부분의 정밀 분석 필요성이 커지고 있다.
연구팀이 새로 개발한 3AIM-seq은 자체 제작한 표준 체외 전사 mRNA(IVT mRNA) 및 합성 DNA 물질을 활용, poly(A) 꼬리의 길이와 구조, 변형 부위를 정밀하게 시퀀싱한다. 분석 과정에는 차세대염기서열 일루미나 플랫폼과 자체 개발한 생명정보학 알고리즘을 적용했다. 연구진은 PCR 증폭 과정에서의 오차를 최소화해, poly(A) 꼬리 길이 예측 정밀도를 ±5 뉴클레오타이드(nt) 수준까지 끌어올렸다고 설명했다.
박대찬 교수는 “이번 연구가 그동안 분석이 까다로웠던 poly(A) 꼬리의 정확한 품질평가에 혁신을 가져다줄 것”이라며 “우리 연구팀이 개발한 분석법은 향후 mRNA 백신과 항암제, 유전자질환 치료제 등 다양한 mRNA 플랫폼 치료제의 국제 표준 도구로 쓰일 수 있다”고 말했다.
이번 연구 결과는 지난달 28일 국제 학술지 '분자 치료'(Molecular Therapy) 온라인판에 게재됐다.
수원=김동성 기자 estar@etnews.com
[Copyright © 전자신문. 무단전재-재배포금지]
이 기사의 카테고리는 언론사의 분류를 따릅니다.
기사가 속한 카테고리는 언론사가 분류합니다.
언론사는 한 기사를 두 개 이상의 카테고리로 분류할 수 있습니다.
언론사는 한 기사를 두 개 이상의 카테고리로 분류할 수 있습니다.


